熱線:021-66110810,66110819
手機:13564362870


熱線:021-66110810,66110819
手機:13564362870
材料與方法
測試沉積物和植物
沉積物樣品采自中國湖北省梁子湖(114°38'23"N, 30°14'28"E)。沉積物基本特征為:含水量72.5±1.3%,有機質(OM)8.85±0.86 g kg?1,總氮0.50±0.06 g kg?1,總磷0.45±0.05 g kg?1。孔隙水的化學組成為:銨態氮(NH?-N)4.39±1.29 mg l?1,硝態氮(NO?-N)0.22±0.07 mg l?1,pH 6.99±0.08。選擇菹草(Potamogeton crispus, Potmogetonaceae),一種常見的多年生草本沉水植物,作為本實驗的寄主植物。這種植物也是中國南方湖泊的本地種。
根箱設計
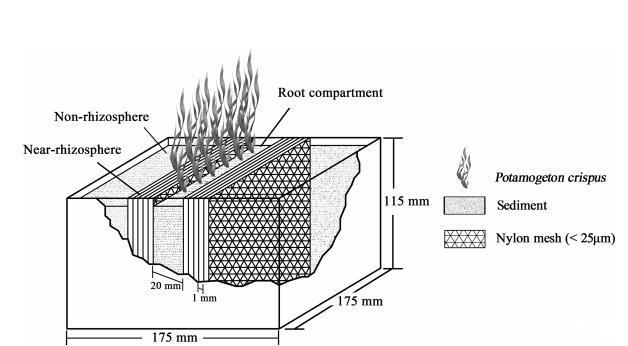
使用具有多層隔室的三隔室根箱培養菹草。根箱根據He et al.(2005)的設計進行了修改(圖1)。每株植物在培養期間完全浸入水箱(高度350毫米)底部。使用風干沉積物填充三個隔室。根箱包含三個部分:根區隔室(寬度20毫米)、近根際區隔室(5毫米)和非根際區隔室(>5毫米)。近根際區隔室通過尼龍網(<25μm)進一步分成五個子隔室(厚度1毫米),以防止根毛進入相鄰的沉積物夾層,并保持沉積物夾層中的微生物和根系分泌物分離。
植物培養與樣品采集
選擇六顆具有相似萌發時間和生長特性的菹草鱗莖種植在根區隔室。植物從2014年11月至2015年5月培養6個月。為了模擬淡水湖泊的低、中、富營養三種營養狀況,使用了緩釋尿素(魯西化工有限公司,聊城,山東,中國)作為營養輸入,濃度分別為0、400和600毫克尿素每千克沉積物。每個營養濃度實驗包含三個重復根箱。培養5個月后,植物根系填滿根區隔室,將緩釋尿素注入沉積物。在尿素注入后第0天開始,每隔14天采集樣品,持續56天,采集根區(R)、五個近根際區子隔室(N1-N5)和非根際區隔室(Non)的沉積物樣品(每種營養條件下三個平行樣品)。來自同一夾層的沉積物樣品混合,然后使用七個混合樣品進行DNA提取、克隆和測序,以檢測每個夾層是否存在類產氧甲烷桿菌。
理化指標測定和基因組DNA提取
在收集沉積物之前,使用微電極系統(Unisense, Aarhus, Denmark)原位測量根際沉積物的溶解氧(DO)和pH。由于微電極系統探針長度有限,測量深度為48毫米。根據測量的DO值確定每個近根際子隔室的氧滲透深度(Dop, DO > 6.25 μmol l?1)。收集的沉積物在4,000 rpm下離心10分鐘以獲得孔隙水。使用流動注射分析(SEAL Analytical AA3; SEAL Analytical, Norderstedt, Germany)測定孔隙水中NH?-N和NO?-N的濃度。
使用土壤快速DNA提取試劑盒(Fast DNA Spin Kit for Soil, MP Biomedicals, Fountain Parkway Solon, OH, USA)按照制造商的說明提取沉積物的基因組DNA。每次DNA分離使用約0.5克沉積物。提取的DNA隨后儲存在-20°C直至進一步分析。使用NanoDrop 2000紫外-可見分光光度計(Thermo Fisher Scientific, Waltham, MA, USA)測定DNA濃度,并通過1.2%瓊脂糖凝膠電泳檢查質量。
擴增、克隆和測序
采用巢式PCR擴增類產氧甲烷桿菌的16S rRNA基因。第一步使用引物202F (5'-GACCAAAGGGGGCGAGCG-3') (Ettwig et al., 2009) 和1545R (5'-CAKAAAGGAGGTGATCC-3') (Juretschko et al., 1998),第二步使用針對NC10細菌的特異性引物qP1F (5'-GGGCTTGACATCCCACGAACCTG-3') 和 qP2R (5'-CGCCTTCCTCCAGCTTGACGC-3') (Ettwig et al., 2009)。反應體系為:12.5 μl 2× High-Fidelity Master Mix (blue), 1 μl 正向引物 (10 μM), 1 μl 反向引物 (10 μM), 1 μl DNA模板 (20-50 ng μl?1), 和 9.5 μl ddH?O。PCR條件為:98°C預變性5分鐘;然后98°C變性10秒、56°C退火10秒、72°C延伸20秒,共35個循環;最后72°C延伸5分鐘。使用pClone 007載體連接試劑盒(TSING KE, 北京, 中國)對PCR產物進行克隆。從每個夾層隨機挑選約50個陽性克隆進行測序以構建克隆文庫。
統計分析
使用方差分析(最小顯著性差異法(LSD)和Tukey檢驗)檢測沉積物和孔隙水理化指標之間的顯著差異。應用3%的操作分類單元(OTU)截斷值來確定類產氧甲烷桿菌16S rRNA基因的遺傳多樣性,并使用Mothur程序(v.1.34.4)(Schloss et al., 2009)中的最遠鄰法(furthest neighbor method)進行序列聚類。使用ClustalW 1.6程序進行多序列比對。使用Mega 6軟件,采用鄰接法(neighbor-joining method)進行16S rRNA基因序列的系統發育分析。計算模型為Jukes-Cantor模型。使用1000次重復的Bootstrap分析來檢驗樹狀圖聚類的置信水平(Shen et al., 2014a)。使用Mothur軟件(v.1.34.4)生成Chao1估計量和香農指數以評估多樣性。本研究獲得的序列在NCBI數據庫中與產氧甲烷桿菌序列(FP565575)進行BLAST以獲取相似性,并在GenBank中上傳,登錄號為MH092300-MH092623。
 相關新聞
相關新聞